
私たちのグループでは、真核生物の遺伝子発現、DNA複製や、それらを制御するクロマチン構造変化といった生命の根幹に関わる謎に挑んでいます。特徴は、世界最先端の1分子イメージング技術と、スーパーコンピュータ等を駆使した分子シミュレーションを融合させた独自のアプローチです。実験、シミュレーション、機械学習を駆使して、クロマチン上で起こる転写、複製、修復といった生命現象を支える分子レベルのダイナミクスを明らかにし、エピジェネティクスや細胞運命制御の新たな理解に貢献することを目指しています。私たちは、新しいメンバーを歓迎しています。分子レベルでこれらの生命現象を理解したい人、世界最先端の実験・シミュレーション技術を身につけたい人であれば生命科学・物理・化学・情報系など、経歴や専門分野は問いません。興味があればメール(terakawa at biophys.kyoto-u.ac.jp)で連絡してください。
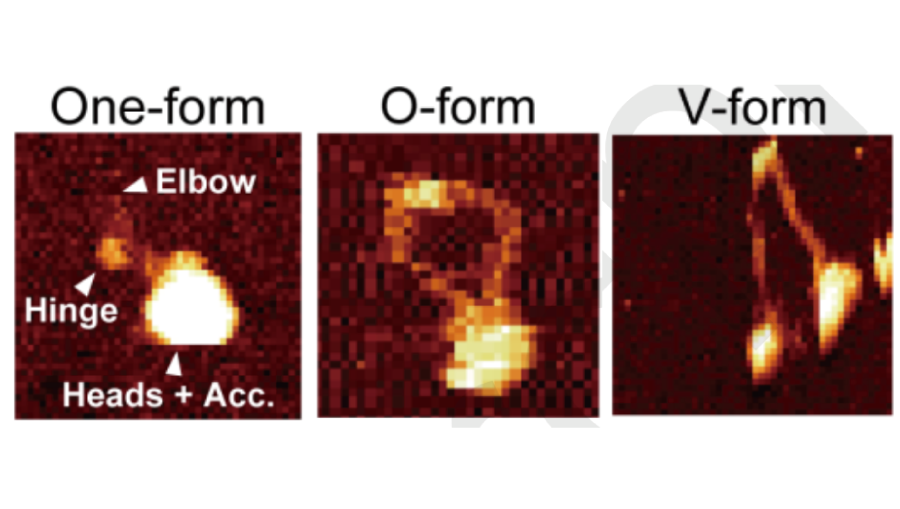
Condensin dynamics
コンデンシンのATP結合と解離に伴う構造変化を、液中AFMと分子動力学シミュレーションで解析しました。その結果、ATP結合によるヘッドの会合がヒンジの開閉と連動し、ADP解離後にBrn1が再結合することが示唆されました。金沢大・古寺グループとの共同研究です。

Cohesin DNA Binding
粗視化分子動力学シミュレーションにより、ヒトコヒーシン複合体のDNA結合部位を特定しました。SMC1、SMC3、STAG1、NIPBLにDNA結合パッチがあり、協調してDNAを曲げることが示されました。さらに、STAG1とNIPBLがループ押し出しをサポートすることも明らかになりました。
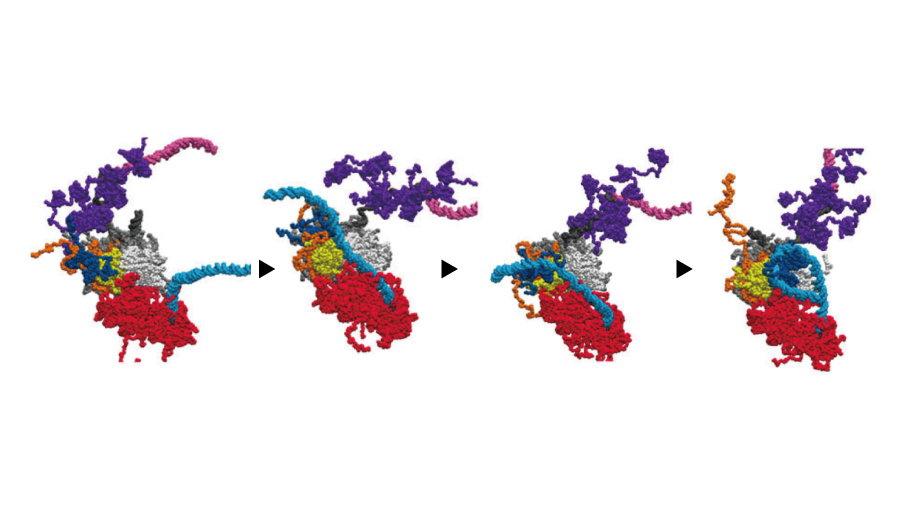
Histone Recycled
粗視化分子動力学シミュレーションにより、DNA複製フォークにおける親由来ヒストンH3/H4のリサイクリング過程を可視化しました。その結果、ヒストンはCdc45依存・非依存の経路を通じて再配置され、RPAとPol εの結合がリサイクリングの鎖選択や位置に影響することがわかりました。
MutS Corkscrewing
粗視化分子動力学シミュレーションを用いて、DNA塩基対ミスマッチ認識タンパク質であるMutSがDNAに沿ってくるくると回りながらスライディングするダイナミクスを可視化しました。その結果、DNAの曲がりに応じてMutSの拡散速度が変化することがわかりました。

Lane Switch on Nucleosome
粗視化分子動力学シミュレーションの結果を、生化学実験とナノポアシーケンシングで検証することで、RNAポリメラーゼや複製ヘリカーゼが、ヌクレオソームをDNAに沿ってスライディングさせることを明らかにしました。その分子機構として「レーン・スイッチ機構」を提唱しました。

Condensin is a Motor
DNAカーテンデバイスを用いた1分子蛍光イメージングによって染色体の高次構造形成に関わるコンデンシンタンパク質が分子モーターであることを明らかにしました。研究の成果はScience誌に掲載され、表紙を飾りました。引き続き、モーターとして働く分子機構を研究しています。
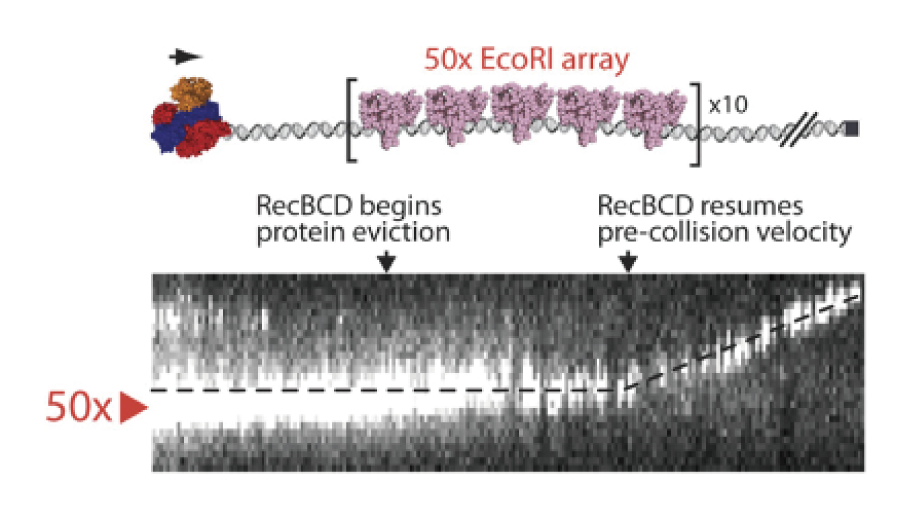
RecBCD Sequential Eviction
1分子蛍光イメージングにより、DNA上に密集して結合したタンパク質群に衝突するRecBCD分子モーターの動態を観察しました。その結果、RecBCDは、個々のタンパク質を順次DNAから剥がしていく独自の機構を用いて、混雑したDNA上でも効率的に移動できることが示されました。
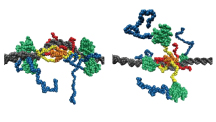
p53 Dancing on DNA
粗視化分子動力学シミュレーションを用いて、がん抑制性転写因子がその認識DNAサイトを探してDNA上をスライディングするダイナミクスを可視化しました。その結果、C末端天然変性領域でDNA上を拡散しながら、DNA結合領域がダンスするように認識サイトを探すことが示唆されました。
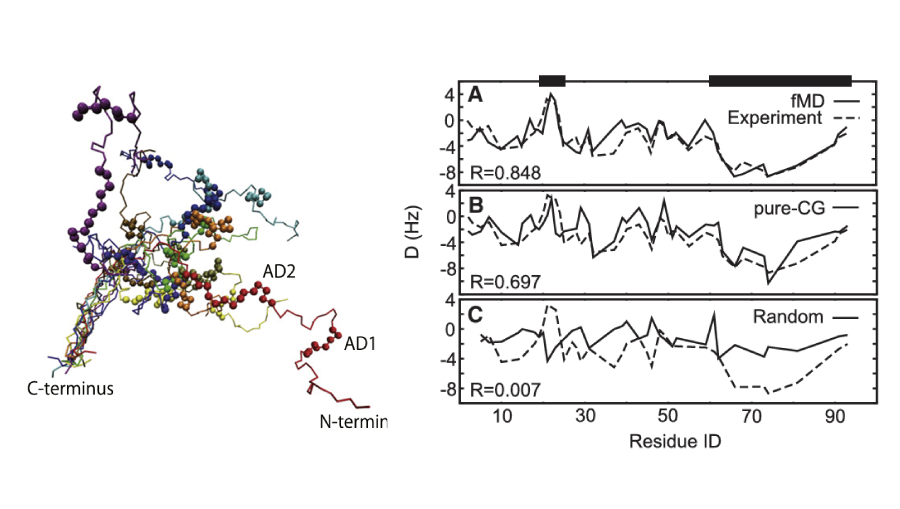
CG for disorder
フラグメントごとの全原子分子動力学シミュレーションと粗視化モデリングを統合したマルチスケールアプローチを開発しました。この手法により、広範な構造アンサンブルを高精度に再現することが可能となりました。得られた構造アンサンブルは、NMRなどの実験データとも一致しました。
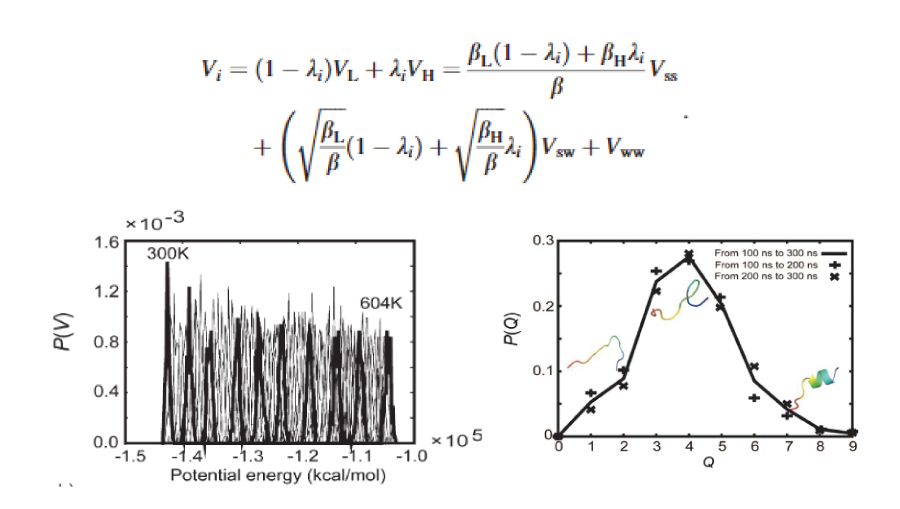
Solute Tempering
REST(Replica Exchange with Solute Tempering)法の変法が、力場パラメータのスケーリングによってGROMACS上で容易に実装できることを示しました。さらに、この手法は、従来のREM法と同等のサンプリング効率を、より少ないレプリカ数で実現できることを確認しました。








